Supponiamo che tu abbia dati di serie temporali con righe e colonne che desideri alimentare a un livello in Keras. Prima di inviarlo a RNN, è necessario rimodellare i dati precedenti in un tensore 3D. Quindi diventa un .N700SimpleRNN(200, return_sequence=True)N× 700 × 1
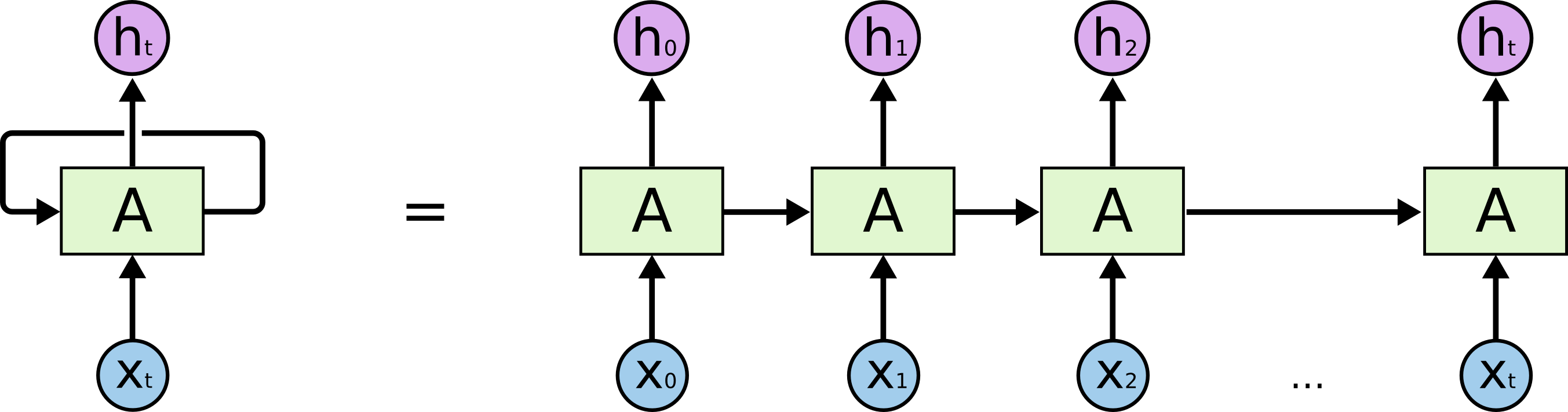
L'immagine è tratta da https://colah.github.io/posts/2015-08-Understanding-LSTMs
In RNN, le colonne (le "700 colonne") rappresentano i timestep di RNN. I tuoi dati vengono elaborati da . Dopo aver fornito i dati a RNN, ora ha 700 output che sono da a , non da a . Ricorda che ora la forma dei tuoi dati è che è campioni (le righe) x timesteps (le colonne) x canali .t = 1 t o 700 h1h700h1h200N× 700 × 200
Quindi, quando applichi a TimeDistributedDense, stai applicando un Denselivello su ogni timestep, il che significa che stai applicando un Denselivello su ogni , , ..., rispettivamente. Il che significa: in realtà stai applicando l'operazione completamente connessa su ciascuno dei suoi canali (quello "200"), rispettivamente, da a . Il primo " " fino al 700 ° " ".h1h2hth1h7001 × 1 × 2001 × 1 × 200
Perché stiamo facendo questo? Perché non si desidera appiattire l'output RNN.
Perché non appiattire l'uscita RNN? Perché vuoi mantenere separati i valori di ogni timestep.
Perché mantenere separati i valori di ogni timestep? Perché:
- vuoi solo interagire i valori tra il proprio timestep
- non vuoi avere un'interazione casuale tra diversi timestep e canali.
