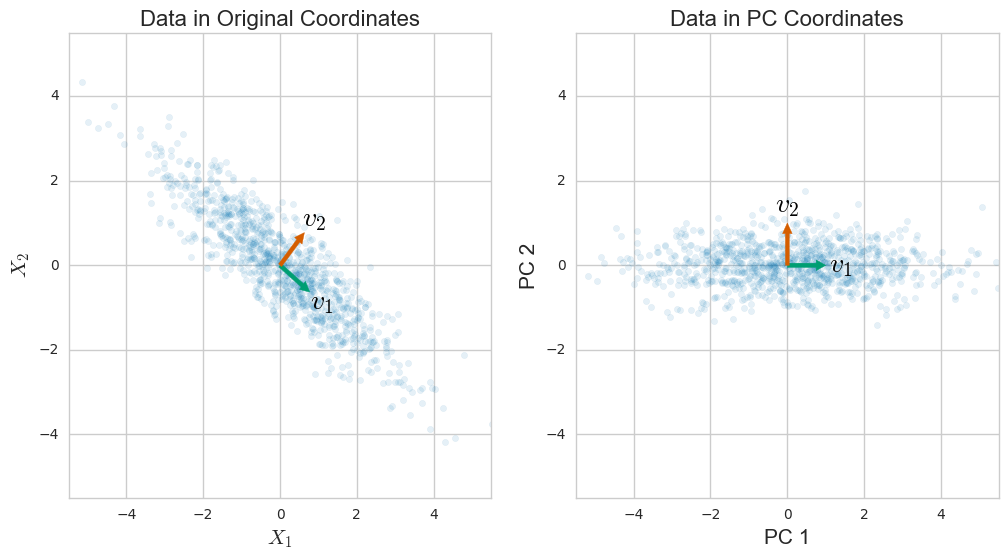
Vorrei iniziare con PCA. Supponiamo di avere n punti dati composti da d numeri (o dimensioni) ciascuno. Se si centrano questi dati (sottraendo il punto di dati medio da ciascun vettore di dati ) è possibile impilare i dati per creare una matriceμxi
X=⎛⎝⎜⎜⎜⎜⎜⎜xT1−μTxT2−μT⋮xTn−μT⎞⎠⎟⎟⎟⎟⎟⎟.
La matrice di covarianza
S=1n−1∑i=1n(xi−μ)(xi−μ)T=1n−1XTX
misure a che livello le diverse coordinate in cui vengono forniti i tuoi dati variano insieme. Quindi, forse non sorprende che PCA - che è progettato per catturare la variazione dei tuoi dati - possa essere dato in termini di matrice di covarianza. In particolare, la decomposizione degli autovalori di risulta essereS
S=VΛVT=∑i=1rλivivTi,
dove è l' -esimo componente principale , o PC, e è l' -esimo valore di ed è anche uguale alla varianza dei dati lungo l' -esimo PC. Questa decomposizione proviene da un teorema generale dell'algebra lineare e si deve fare del lavoro per motivare il relatino alla PCA.viiλiiSi
SVD è un modo generale per comprendere una matrice in termini di spazio colonna e spazio riga. (È un modo per riscrivere qualsiasi matrice in termini di altre matrici con una relazione intuitiva con lo spazio di riga e colonna.) Ad esempio, per la matrice possiamo trovare le indicazioni e nel dominio e spaziare in modo taleA=(1021)uivi
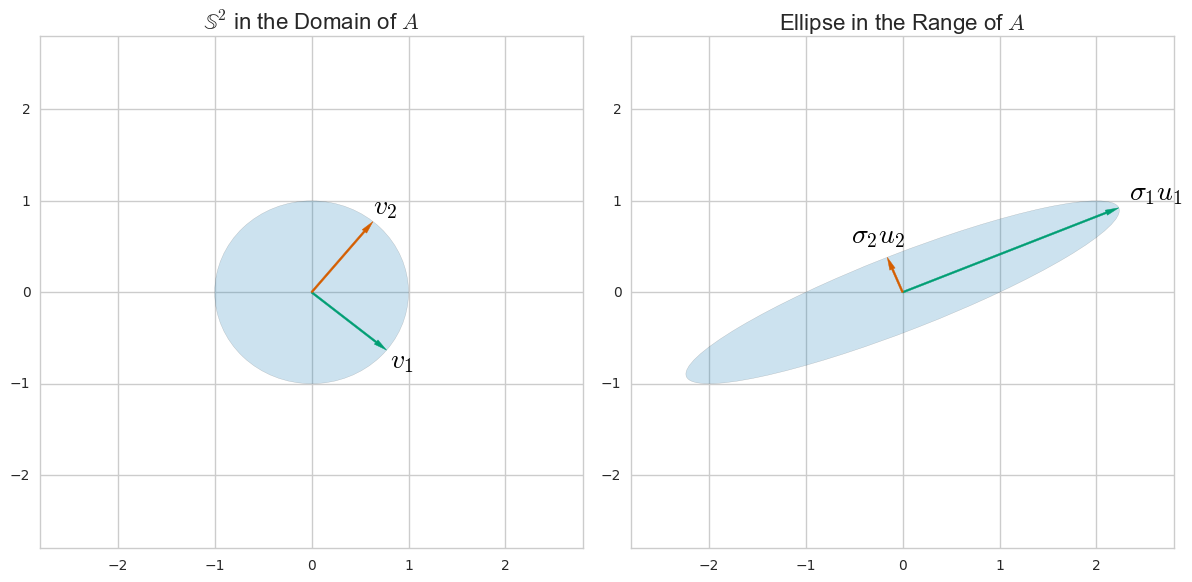
Puoi trovarli considerando come come trasformazione lineare trasforma una sfera unitaria nel suo dominio in un'ellisse: i semiassi principali dell'ellisse si allineano con e sono le loro preimmagini.ASuivi
In ogni caso, per la matrice di dati sopra (davvero, basta impostare ), SVD ci permette di scrivereXA=X
X=∑i=1rσiuivTj,
dove e sono insiemi di vettori ortonormali. Un confronto con la decomposizione degli autovalori di rivela che i "vettori singolari giusti" sono uguali ai PC, i "vettori singolari giusti" sono{ v i } S v i{ui}{vi}Svi
ui=1(n−1)λi−−−−−−−−√Xvi,
e i "valori singolari" sono correlati alla matrice di dati tramiteσi
σ2i=(n−1)λi.
E 'un fatto generale che i vettori singolari giusti coprono lo spazio di colonna di . In questo caso specifico, ci fornisce una proiezione in scala dei dati sulla direzione -componente principale. I vettori singolari di sinistra in generale si estendono nello spazio di riga di , il che ci fornisce un insieme di vettori ortogonali che coprono i dati in modo molto simile ai PC. X u i X i v i XuiXuiXiviX
Vado in ulteriori dettagli e vantaggi della relazione tra PCA e SVD in questo articolo più lungo .


